KINASCREENTM
Charakterisierung der Bindungseigenschaften von Kinasen mit kleinen Molekülen durch SPR basierte Assays
Nutzen Sie unseren KINASCREENTM Service um die Bindungscharakterstika kleiner Moleküler und Kinasen zu untersuchen.
Wir haben bereits Bindungsassays für mehr als 25 Kinasen etabliert zur schnellen und umfangreichen Charakterisierung der kinetischen Bindungseigenschaften von Kinase-Inhibitoren und deren Target-Enzym. Dabei wird die Kinase als Ligand auf dem Sensorchip immobilisiert, das zu untersuchende Molekül über die Oberfläche geleitet und deren Wechselwirkung in Echtzeit detektiert. Biacore - Bindungsassays sind für folgende Kinasen bereits verfügbar:
- EGF-R
- ERBB4
- ERK1 (MAPK3)
- FGF-R1
- JNK2 (MAPK9)
- LYN
- c-MET
- p38alpha (MAPK14)
- p38beta (MAPK11)
- p38gamma (MAPK12)
- p38delta (MAPK13)
- PKA (catalytic and regulatory subunits)
- PKB/AKT
- SRC
- YES
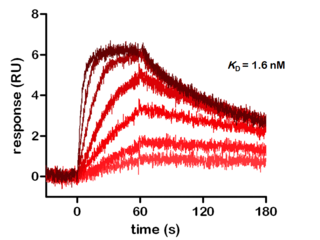
Figure: Kinetische Analyse des universellen kinase Inhibitor Staurosporine mit Bindung an PKA Cα (SPR-Assay).
Unser Service:
- Immobilisierung von aktiver und / oder inaktiver Kinase mittels verschiedener Kopplungsstrategien
- Qualitatives Screening oder Semi-qualitative Analyse von Kinaseinhibitoren (kleine Moleküle)
- Quantitative kinetische Analyse der Bindung (kinetische Konstanten, Affinitätskonstante kass, kdiss, KD)
- Thermodynamische Charakterisierung der biomolekularen Wechselwirkung, Bestimmung der thermodynamischen Konstanten des Gleichgewichts- und des Übergangszustandes
Ihr Targetenzym ist nicht dabei ? Wir arbeiten stetig daran weitere SPR - basierte Assays für Kinasen anzubieten. Bitte kontaktieren Sie einen unsere Applikationsspezialisten um weiterführende, detaillierte Informationen zu erhalten. Gerne erstellen wir Ihnen auch ein individuelles Angebot.
Wir schätzen Ihre Privatsphäre
Um unsere Webseite für Sie optimal zu gestalten und fortlaufend verbessern zu können, verwenden wir Cookies. Weitere Informationen erhalten Sie in unserer Datenschutzerklärung. Hier finden Sie unser Impressum.